El cáncer es una de las principales causas de muerte en todo el mundo. En México, el cáncer de mama representa la primera causa de muerte en mujeres mexicanas con cáncer y se estima que, en los siguientes veinte años, habrá un incremento del 50% en la mortalidad e incidencia de este cáncer en America Latina (Rowley, J. et. al. 2015).
Además del cáncer de mama, el cáncer gástrico representa una de las neoplasias malignas más comunes en México, con aproximadamente 7500 casos nuevos registrados cada año. El cáncer gástrico es una de las malignidades más letales, con un número estimado de 6000 casos anuales y una razón de mortalidad/incidencia de 0.8. A pesar de que ambas malignidades son muy frecuentes en México, se sabe muy poco en cuanto al paisaje genómico de las células tumorales y el microambiente celular circundante incluyendo el infiltrado inmune en tumores de ambos tipos en pacientes mexicanas y mexicanos.
La biología y el origen del cáncer es compleja y la heterogeneidad que presenta en cuanto a los tipos celulares, mutaciones y re-arreglos cromosómicos que llevan al desarrollo de la enfermedad representa uno de los grandes retos en términos de su diagnóstico y el tratamiento eficiente (Wu, S. et. al. 2021).
En este proyecto identificaremos los mecanismos moleculares y componentes celulares de origen inmune y estromal asociados con la malignidad tumoral de cáncer de mama y cáncer gástrico por medio de la caracterización a nivel transcriptómico de células individuales del tumor en contraste con tejido sano adyacente lo cual nos permitirá recapitular la diversidad celular que componen al tumor y al nicho tumoral incluyendo el aporte inmune.
Existen evidencias convincentes que sugieren que subtipos celulares inmunes y citocinas efectoras son clave en la supresión del crecimiento tumoral mientras que otras son nocivas, ya que colaboran con las células tumorales para evadir a la respuesta inmune. El conocimiento adquirido de este fenómeno a la fecha, ha colocado a las inmunoterapias como una de las alternativas más prometedoras para tratar y curar el cáncer.
Así mismo caracterizaremos los tumores a nivel genómico-estructural por medio de tecnologías de reconstrucción de la topología genómica, para así identificar las alteraciones cromosómicas y las aberraciones en la organización genómica presentes en el tumor en contraste con el tejido sano (Dixon, J. et. al. 2018).
La integración de los datos transcriptómicos con los topológicos nos permitirán detallar los mecanismos a nivel molecular que conllevan a la expresión aberrante de genes contribuyendo a la transformación celular y de esta forma proponer nuevos candidatos moleculares como biomarcadores y así sentar las bases del conocimiento necesario para, en un futuro, desarrollar herramientas terapéuticas y diagnósticas personalizadas más eficaces.
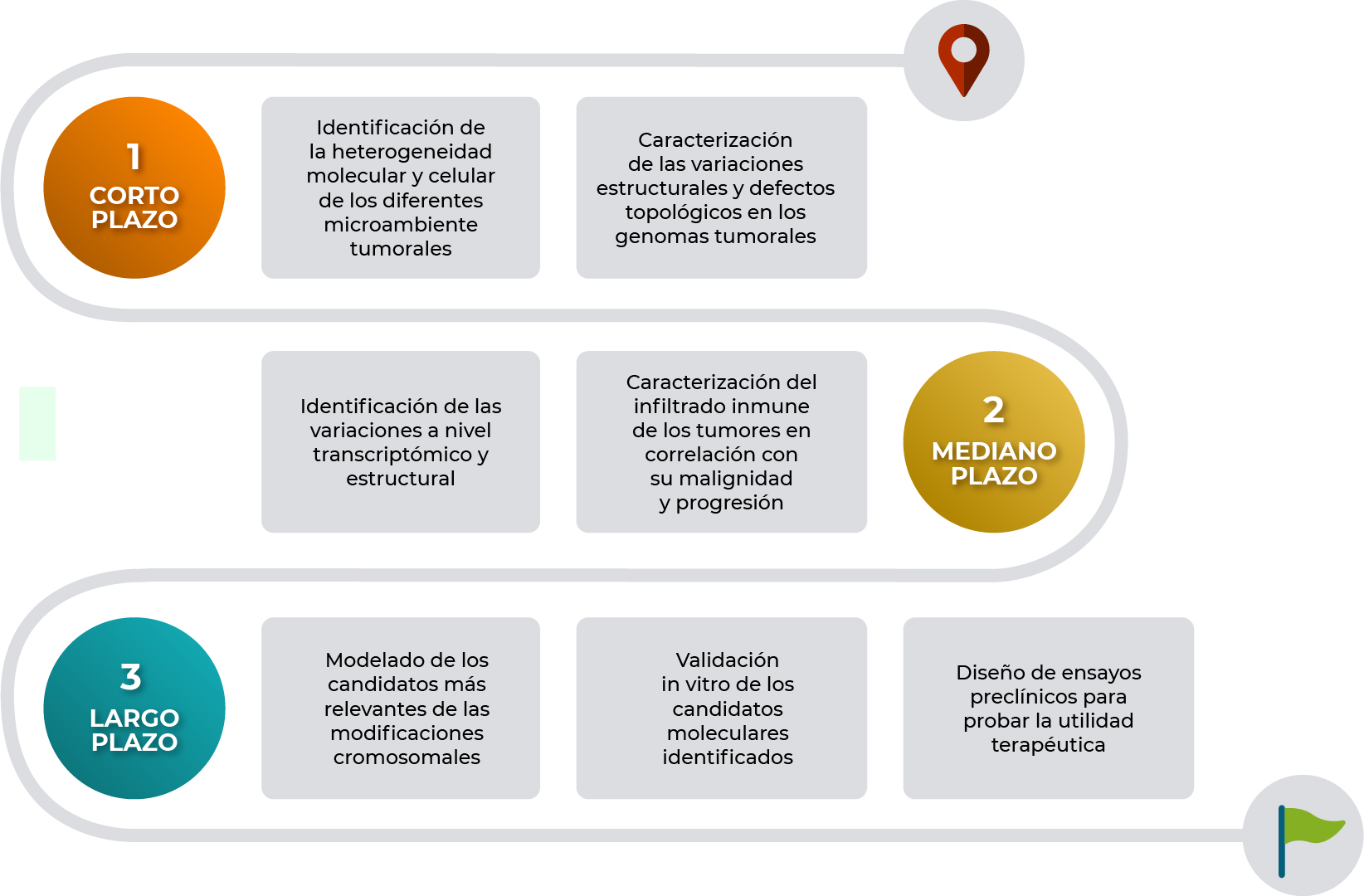
La metodología general de este proyecto consiste en diferentes fases:
- Colecta de muestras (tumor, tejido adyacente no tumoral, sangre periférica) de pacientes con cáncer de mama y cáncer gástrico previo consentimiento informado del protocolo (n=20).
- Procesamiento de muestras para generar una suspensión de células individuales con alta viabilidad.
- Obtención de transcriptomas de células individuales utilizando la tecnología de 10X Genomics.
- Aplicación de captura conformacional de cromosomas de todo el genoma (Hi-C) para obtener la estructura 3D del genoma y los rearreglos cromosomales.
- Análisis de los transcriptomas de células individuales
- Análisis de la estructura genómica y rearreglos cromosomales
- Integración de datos y anotación de rearreglos cromosomales
El proyecto genera entregables a lo largo de las diferentes etapas de desarrollo:
REFERENCIAS
J. D. Rowley, M. M. Le Beau, and T. H. Rabbitts et. al. Chromosomal Translocations and Genome Rearrangements in Cancer. (2015). Springer Cham. https://link.springer.com/book/10.1007/978-3-319-19983-2
Wu, S.Z., Al-Eryani, G., Roden, D.L. et. al. (2021). A single-cell and spatially resolved atlas of human breast cancers. Nature Genetics. 53, 1334–1347. https://doi.org/10.1038/s41588-021-00911-1
Dixon, Jesse R., Jie Xu, Vishnu Dileep, Ye Zhan, Fan Song, Victoria T. Le, Galip Gürkan Yardımcı, Abhijit Chakraborty. (2018).«Integrative Detection and Analysis of Structural Variation in Cancer Genomes». Nature Genetics. 50(10):1388-98. https://doi.org/10.1038/s41588-018-0195-8

Responsable
Dra. Mayra Furlan Magaril
Investigadora Titular A
Instituto de Fisiología Celular
Universidad Nacional Autónoma de México (UNAM)
Colaboradores
- Dr. Enrique Soto Pérez de Celis
Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán
INCMNSZ - Dra. Yanin Chávarri Guerra
Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán
INCMNSZ - Lic. Andrea Morales Alfaro
Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán
INCMNSZ - Dr. Armando Gamboa Domínguez
Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán
INCMNSZ - Dr. Rubén Cortés González
Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán
INCMNSZ - Dr. Alejandro Alfaro Goldaracena
Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán
INCMNSZ - Dr. Guido Grajales Figueroa
Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán
INCMNSZ - Dr. Isaac Bartnicki Navarrete
Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán
INCMNSZ - Patólogo Fernando Candanedo González
Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán
INCMNSZ
- Penagos-Puig Andrés, Claudio-Galeana Sherlyn, Stephenson-Gussinye Aura, Jácome-López Karina, Aguilar-Lomas Amaury, Pérez-Molina Rosario, Furlan-Magaril Mayra. RNA polymerase II pausing contributes to maintain chromatin organization in erythrocytes. bioRxiv 2022.06.16.496295; doi: https://doi.org/10.1101/2022.06.16.496295
- Delgado-Olguín P, Oktaba K and Furlan-Magaril M (2021) Editorial: Chromatin Spatial Configuration and Function in Metazoans. Front. Genet. 12:734981. https://doi.org/10.3389/fgene.2021.734981
- Esquivel-López, A., Arzate-Mejía, R., Pérez-Molina, R., Furlan-Magaril, M. In Nucleus Hi-C in Drosophila Cells. J. Vis. Exp. (175), e62106. https://www.jove.com/es/t/62106